Etapas da tradução
Introdução
Já se perguntou de que maneira os antibióticos matam as bactérias—por exemplo, quando você tem uma sinusite? Diferentes antibióticos atuam de maneiras diferentes, mas alguns atacam um processo bem básico nas células bacterianas: eles eliminam a capacidade de produzir novas proteínas.
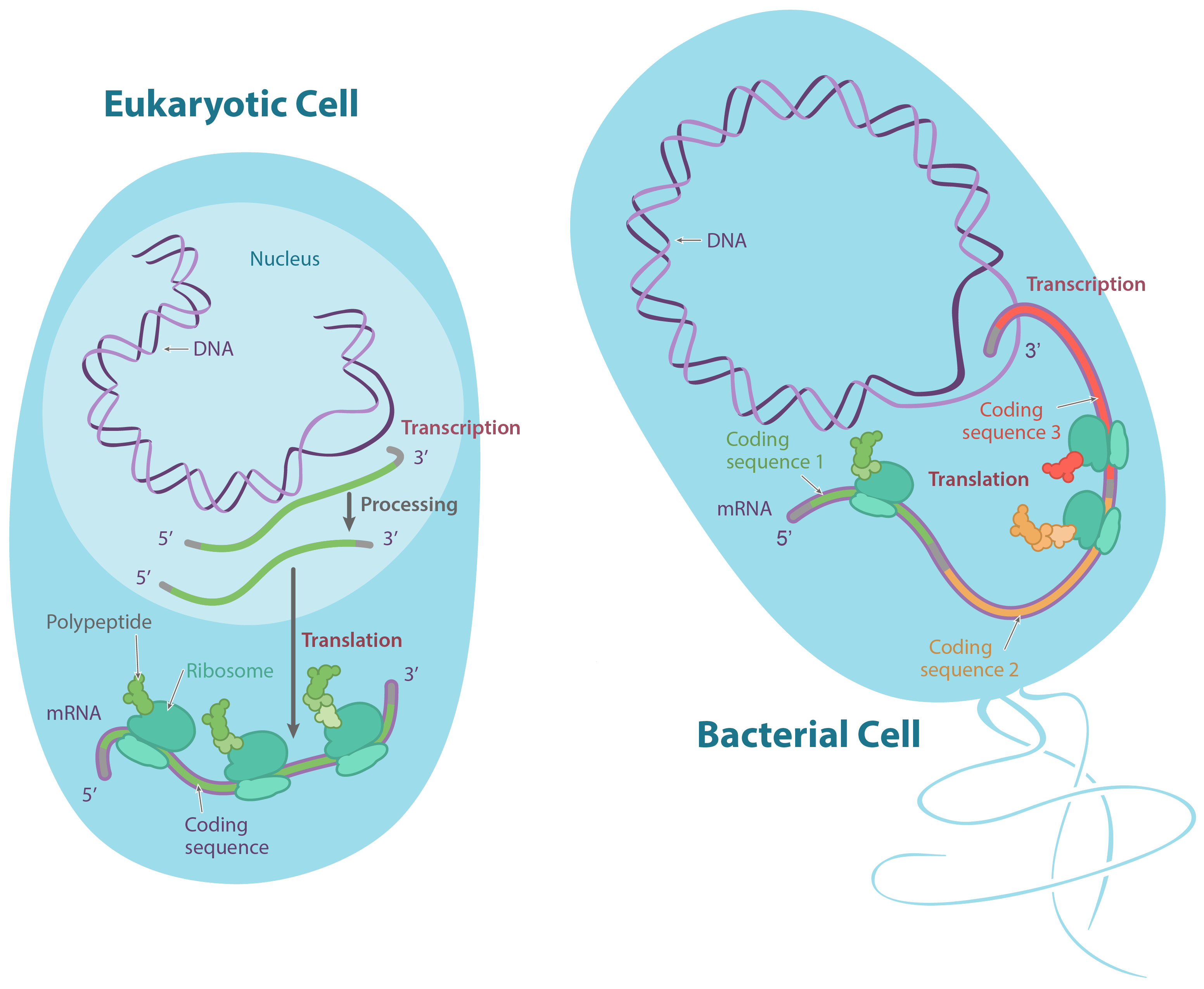
Para usarmos um pouco do vocabulário da biologia molecular, esses antibióticos bloqueiam a tradução. No processo de tradução, a célula lê a informação de uma molécula chamada RNA mensageiro (RNAm) e usa essa informação para construir uma proteína. A tradução acontece constantemente em uma célula bacteriana normal, assim como na maioria das células de seu corpo e é fundamental para mantê-lo vivo (e suas bactérias "visitantes" também).
Quando você toma certos antibióticos (por exemplo, eritromicina), a molécula de antibiótico se associa a moléculas fundamentais para a tradução de dentro da célula bacteriana e basicamente as paralisa. Sem uma maneira de produzir proteína, a bactéria para de funcionar e vai morrer eventualmente. Por esse motivo que infecções são curadas quando tratadas com antibióticos.start superscript, 1, comma, 2, end superscript
As células precisam da tradução para se manterem vivas , e entender o funcionamento (para que possamos impedi-la com antibióticos) pode nos salvar das infecções bacterianas. Vamos analisar como acontece a tradução, da primeira etapa até o produto final.
Tradução: visão geral
A tradução envolve "decodificar" um RNA mensageiro (RNAm) e usar sua informação para produzir um polipeptídeo ou cadeia de aminoácidos. No geral, polipeptídeo é basicamente uma proteína (com a diferença técnica que algumas proteínas grandes são formadas por muitas cadeias de polipeptídeos.
O código genético
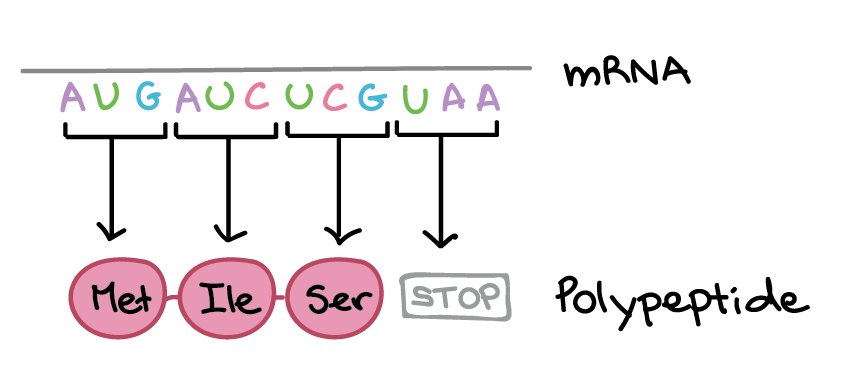
Em um RNAm, as instruções para a produção de um polipeptídeo vêm em grupos de três nucleotídeos chamados códons. Aqui estão algumas características principais dos códons para termos em mente ao seguirmos a diante:
- Existem 61 códons diferentes para os aminoácidos
- Três códons de "parada" marcam o polipeptídeo como terminado
- Um códon AUG é um sinal de "início" para começar a tradução (também especifica o aminoácido metionina)
Essas relações entre os códons do RNAm e os aminoácidos são conhecidas como código genético (sobre o qual você pode explorar mais no artigo código genético.
De códons a aminoácidos
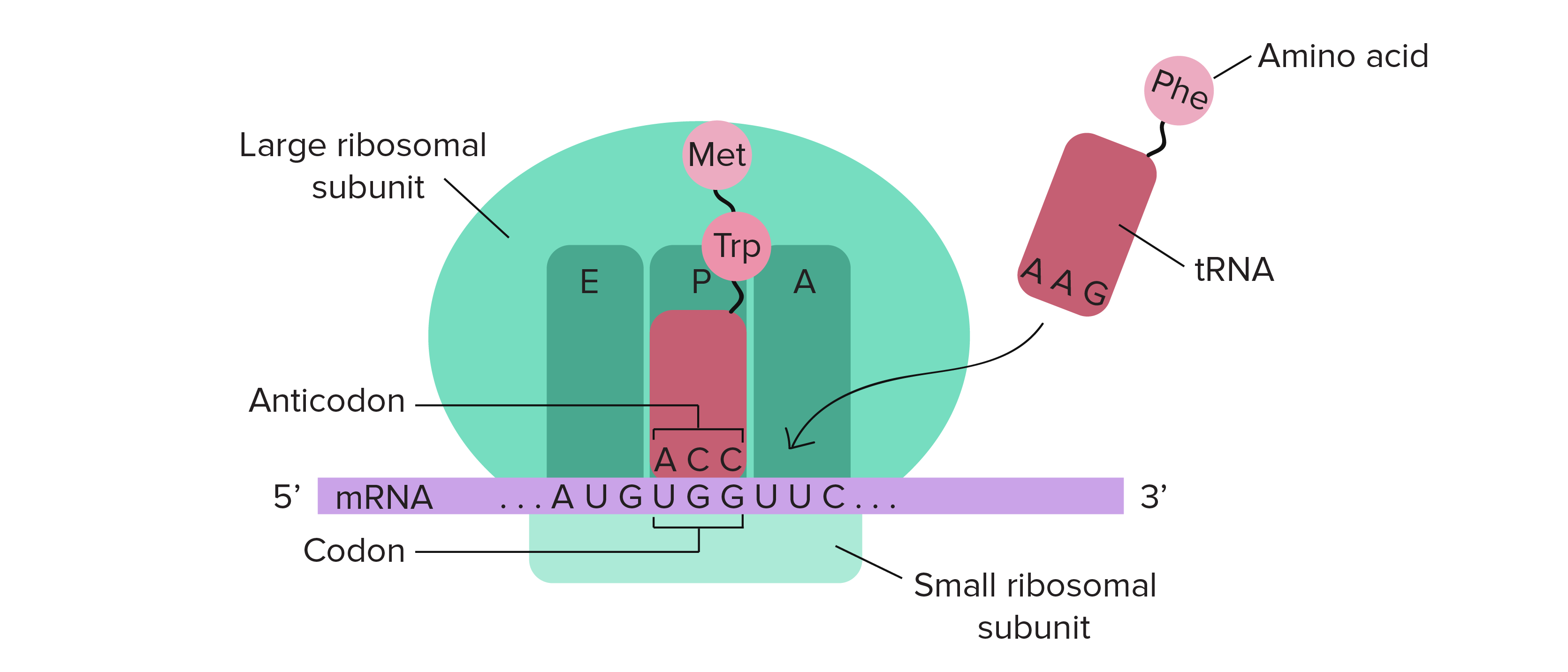
Na tradução, os códons de um RNAm são lidos por ordem (da extremidade 5' para a extremidade 3') por moléculas chamadas de RNAs de transferência ou RNAt.
Cada RNAt possui um anticódon, um conjunto de três nucleotídeos que se liga ao códon correspondente no RNAm através do pareamento de bases. A outra extremidade do RNAt traz o aminoácido especificado pelo códon.
Os RNAt se ligam aos RNAm dentro de uma estrutura de RNA e proteína chamada ribossomo. À medida que os RNAt preenchem os compartimentos do ribossomo e se ligam aos códons, seus aminoácidos são adicionados à cadeia crescente de polipeptídeos em uma reação química. O produto final é um polipeptídeo cuja sequência de aminoácidos reflete a sequência de códons no RNAm.
Essa é a visão geral da tradução. E quanto aos pormenores de como a tradução se inicia, age e termina? Vamos ver.
Tradução: começo, meio e fim
Um livro ou um filme possuem três partes básicas: um começo, um meio e um fim. A tradução tem praticamente estas mesmas três partes, mas com nomes mais elegantes: iniciação, alongamento e terminação.
- Iniciação ("começo"): nesta etapa, o ribossomo se junta ao RNAm e ao primeiro RNAt para que a tradução possa ter início.
- Alongamento ("meio"): nesta etapa, os aminoácidos são trazidos ao ribossomo pelos RNAt e são ligados entre si para formar uma cadeia.
- Terminação ("fim"): na última etapa, o polipeptídeo final é liberado para que possa cumprir sua função na célula.
Vamos analisar como funciona cada etapa.
Iniciação
Para que se possa dar início à tradução, alguns ingredientes são necessários. Esses incluem:
- Um ribossomo (o qual tem duas parte, uma pequena e uma grande)
- Um RNAm com as instruções para a proteína que será construída
- Um RNAt "iniciador" transportando o primeiro aminoácido da proteína, que quase sempre é a metionina (Met)
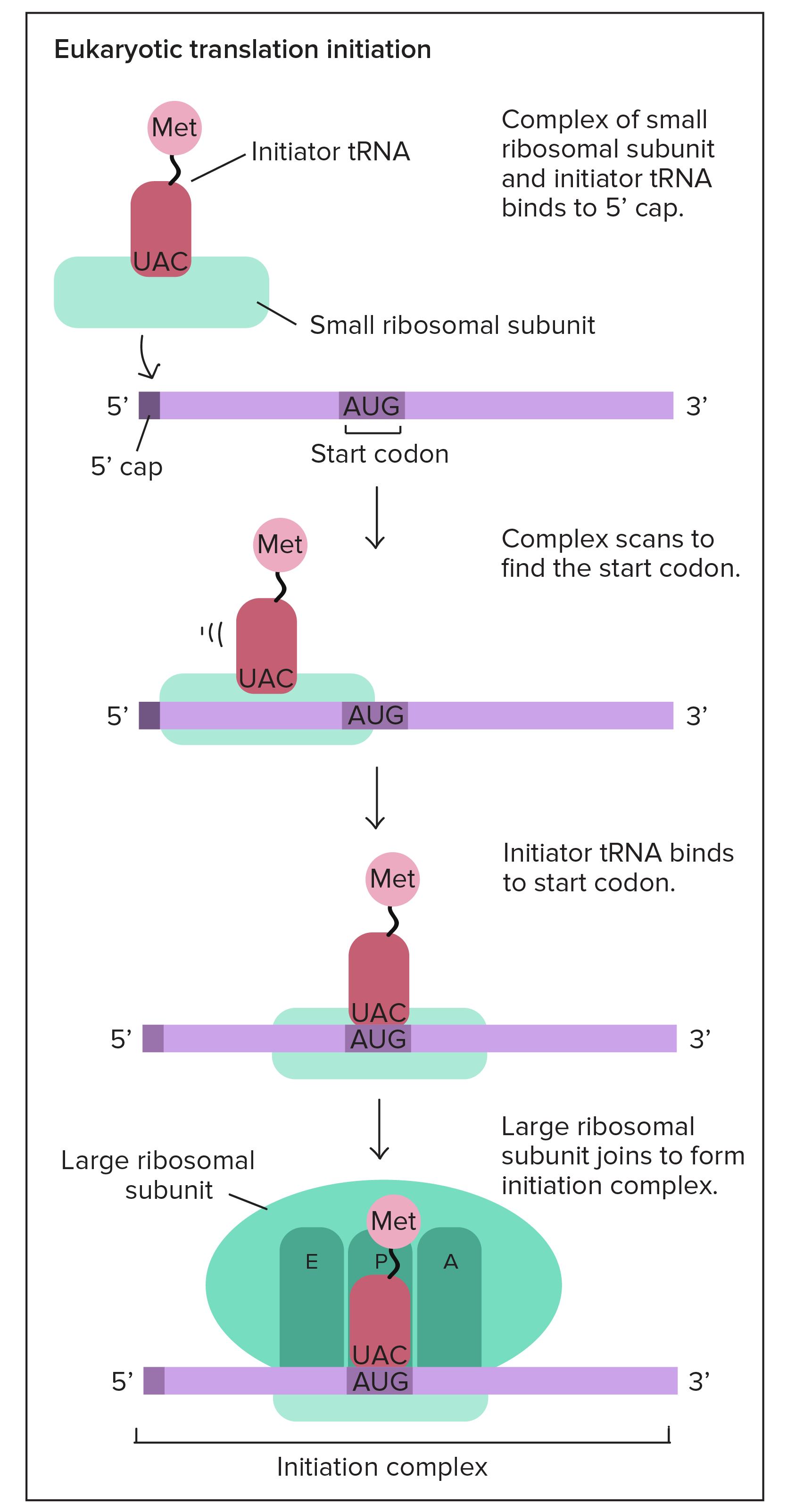
Durante a iniciação, essas peças devem se unir de maneira correta. Juntas, elas formam o complexo de iniciação, a configuração molecular necessária para começar a fazer uma nova proteína.
Dentro de suas células (e das células de outros eucariontes), a iniciação da tradução acontece assim: primeiro o RNAt transportando a metionina se liga a subunidade ribossômica pequena. Juntos, se ligam à extremidade 5' do RNAm através do reconhecimento do cap 5' GTP (adicionado durante o processamento no núcleo). Em seguida, eles "caminham" ao longo do RNAm na direção 3' e param quando alcançam o códon de iniciação (com frequência, mas nem sempre, o primeiro AUG).start superscript, 6, end superscript
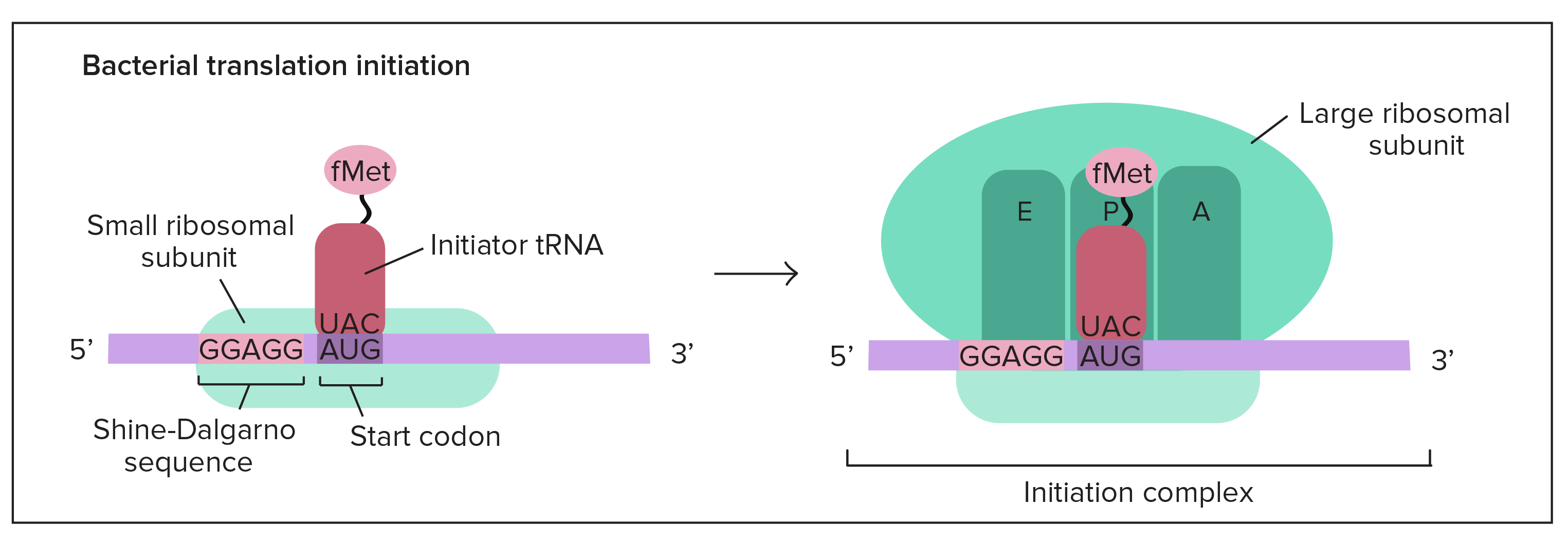
Nas bactérias, a situação é um pouco diferente. Aqui, a subunidade ribossômica pequena não começa pela extremidade 5' e caminha em direção a 3'. Em vez disso, ela se associa diretamente a determinadas sequências no RNAm. Essas sequências Shine-Dalgarno antecedem os códons e "os apontam" para o ribossomo.
Por que usar sequências Shine-Dalgarno? Os genes bacterianos são frequentemente traduzidos em grupos chamados operons), portanto um RNAm bacteriano pode conter as sequências codificadoras de muitos genes. Uma sequência de Shine-Dalgarno marca o início de cada sequência codificadora, o que permite que o ribossomo encontre o códon de iniciação certo para cada gene.
Alongamento
Eu gosto de lembrar o que acontece nessa etapa do "meio" da tradução por seu nome prático: alongamento é quando o polipeptídeo se torna mais longo.
Mas como exatamente a cadeia aumenta? Para descobrirmos, vamos examinar a primeira rodada do alongamento—depois de formado o complexo de iniciação, porém antes que qualquer aminoácido tenha sido adicionado para formar a cadeia.
Nosso primeiro RNAt transportador de metionina começa no compartimento do meio no ribossomo, chamado de sítio P. Ao lado, um novo códon é exposto em outro compartimento, chamado de sítio A. O sítio A será o "desembarque" para o próximo RNAt, aquele cujo anticódon é o correspondente perfeito (complementar) do códon em exposição.
Uma vez que o RNAt correspondente desembarca no sítio A é hora da ação: isto é, a formação de uma ligação peptídica que conecta um aminoácido a outro. Esta etapa transfere a metionina do primeiro RNAt para o aminoácido do segundo RNAt no sítio A.
Nada mal—agora temos dois aminoácidos, um (minúsculo) polipeptídeo! A metionina forma o N-terminal do polipeptídeo, e o outro amioácido é o C-terminal.
Contudo... as maiores chances são que queiramos um polipeptídeo mais longo do que dois aminoácidos. Como a cadeia continua a aumentar? Depois que a ligação peptídica é formada, o RNAm é puxado para frente no ribossomo por exatamente um códon. Esse deslocamento permite que o primeiro RNAt, agora vazio, saia através do sítio E (do inglês "exit"). Isso também faz com que um novo códon fique exposto no sítio A, para que todo ciclo se repita.
E se repete... de alguma vezes até espantosas
33, comma000 vezes! A proteína titina, que é encontrada
em nossos músculos e é o mais longo polipeptídeo conhecido,
pode ter mais de
33, comma000 aminoácidos
start superscript, 8, comma, 9, end superscrip.Terminação
Polipeptídeos, assim como todas as boas coisas, devem eventualmente chegar a um fim. A tradução acaba em um processo chamado terminação. A terminação acontece quando um códon de parada no RNAm (UAA, UAG ou UGA) entra no sítio A.
Códons de parada são reconhecidos por proteínas chamadas de fatores de liberação, os quais se adaptam perfeitamente no sítio P (embora não sejam RNAt). Fatores de liberação confundem a enzima que normalmente forma as ligações peptídicas: fazem-na adicionar uma molécula de água ao último aminoácido da cadeia. Essa reação separa a cadeia do RNAt, assim a proteína recém-produzida é liberada.
E depois? Por sorte, o "equipamento" de tradução é bastante reutilizável. Depois que as unidades ribossômicas se separam do RNAm e entre si, cada elemento pode (e normalmente fazem isso rapidamente) tomar parte em um novo ciclo de tradução.
Epílogo: Processamento
Agora nosso polipeptídeo tem todos os seus aminoácidos—isso significa que esteja pronto para atuar na célula?
Não necessariamente. Polipeptídeos muitas vezes precisam de algumas "edições". Durante e após a tradução, aminoácidos podem ser quimicamente alterados ou removidos. O novo polipeptídeo também vai se dobrar em uma estrutura em 3D distinta e podem se associar a outros polipeptídeos para formar uma proteína com múltiplas partes.
Muitas proteínas são boas em se dobrarem sozinhas, mas algumas precisam de auxiliares ("chaperonas") para evitar que se unam incorretamente durante o processo complexo que é o dobramento.
Algumas proteínas também contêm sequências especiais de aminoácidos que as direcionam para determinadas partes das células. Estas sequências, muitas vezes encontradas próximas aos terminais N e C, podem ser entendidas como as "passagens de trem" das proteínas para suas destinações finais.





0 Comentários